[연구필요성]
패스웨이(Pathway)는 단백질, 유전자, 세포 등 생체 요소간의 상호작용과 역학관계를 세밀하게 설명할 수 있는 생물학적 심층지식을 말한다. 패스웨이 (pathway) 분석은 유전자와 단백질과 같은 생물학적 인자를 기반으로 질병과 관련된 패스웨이를 식별하는 데 사용되어져 왔다. 이러한 질병 연관 패스웨이의 발견은 생물학적 인자들이 어떠한 생물학적 현상을 통해 질병에 영향을 미치는지에 대한 해석에 도움을 주어 향후 질병의 기작 이해에 도움을 줄 수 있다는 장점이 있다.
그러나 기존의 패스웨이 분석 방법론은 대부분 생물학적 인자와 패스웨이 사이의 복잡한 비선형적 관계를 고려하지 못한다는 한계점이 있어 생물학적 복잡성을 반영하지 못하고 있다. 박태성교수 연구팀에서는 이러한 한계점을 개선하기 위하여 딥 러닝을 활용한 모델 DeepHisCoM을 개발하였다. DeepHisCoM은 패스웨이에 대한 생물학적 인자의 복합적이고 비선형적인 기여를 딥 러닝을 활용하여 성공적으로 반영하였으며 동시에 계층적인 생물학적 구조를 반영한 창의적인 통계학적 모델이다.
[연구성과/기대효과]
본 연구에서는 DeepHisCoM을 네 가지 종류의 오믹스 자료와 두 가지 종류의 질병에 대해 적용하여 질병 연관 패스웨이를 찾았으며, 특히 영국의 대규모 UK Biobank의 유전체 SNP 자료를 이용한 COVID-19 환자들의 중증도에 대한 패스웨이 분석을 통해 기존에 COVID-19 연관 패스웨이로 알려진 다수의 패스웨이 및 질병의 이해에 도움을 줄 수 있는 후보 패스웨이를 성공적으로 찾았다. 또한 시뮬레이션 분석을 통하여 기존의 비선형적 관계를 고려하지 않는 모델과 비교하였을 때 DeepHisCoM이 더 높은 성능을 보이는 것을 확인하였다. 이를 통해 본 연구에서는 DeepHisCoM의 다양한 오믹스 자료 및 질병에 대한 적용 가능성을 확인하였으며, 향후 질병 연관 패스웨이 발굴을 통해 질병에 대한 생물학적 기전을 해석하는데 기여할 수 있을 것으로 기대한다.
[본문]
서울대학교 통계학과 박태성 교수는 김보람연구원과 박찬우연구원과 공동으로 딥 러닝에 기반하여 유전자, 단백질 등과 같은 생물학적 인자의 복잡한 비선형적 상호작용을 반영하여 패스웨이 분석을 할 수 있는 방법론 ‘DeepHisCoM’을 개발하였다.
패스웨이 분석은 질병 연관 패스웨이를 찾음으로써 생물학적 인자가 어떠한 생물학적 상호작용, 즉 패스웨이를 통하여 질병에 영향을 미치는지 그 기전을 해석하는데 도움을 줄 수 있다. 기존의 패스웨이 분석 방법론은 생물학적 인자간의 비선형적 관계를 반영하지 못한다는 한계를 가지고 있었다. 박태성교수 연구팀은 이러한 한계를 극복하기 위하여 딥 러닝을 활용한 새로운 패스웨이 방법인 ‘DeepHisCoM’을 개발하였다. ‘DeepHisCoM’은 비선형적 관계를 반영할 뿐 아니라 생물학적 인자에서 패스웨이, 질병으로 흐르는 계층적인 생물학적 구조를 반영하며 동시에 대량의 생물학적 인자를 입력받아 수십, 수백 개의 패스웨이를 동시에 분석할 수 있는 딥 러닝 기반의 새로운 패스웨이 분석 방법이다.
박태성교수 연구팀은 DeepHisCoM의 다양한 생물학적 자료 및 질병에 대한 효용성을 확인하기 위하여 네 가지 종류의 오믹스 자료와 두 가지 종류의 질병에 대해 적용하여 각 질병과 유의미하게 연관된 패스웨이를 찾았다.
영국 UK Biobank의 COVID-19 환자들의 유전체 (SNP) 자료를 이용하여 중증도와 연관된 패스웨이를 분석한 결과 MAPK signaling pathway, GnRH signaling pathway, hypertrophic cardiomyopathy, dilated cardiomyopathy와 그 외 기존 COVID-19와 연관된 것으로 밝혀진 다수의 패스웨이를 찾았다. COVID-19 기전의 이해에 도움을 줄 수 있는 다수의 후보 패스웨이를 찾아 향후 감염병 연구에의 활용 가능성을 제시하였다.
박태성교수 연구팀은 간암 환자의 대사체 자료를 분석하여 lysine degradation, valine, leucine, and isoleucine biosynthesis, phenylalanine, tyrosine, and tryptophan 등 간암 연관 패스웨이를 찾았고, 전사체 및 메타지놈 자료에서 결과의 재현성을 확인하였다. 또한 추가적인 시뮬레이션 분석을 통하여 기존의 비선형적 관계를 고려하지 않는 모델과 비교하였을 때 DeepHisCoM이 더 높은 성능을 보이는 것을 확인하였다.
박태성교수 연구팀은 DeepHisCoM의 다양한 오믹스 자료 및 질병에 대한 적용 가능성을 확인하였으며, 향후 질병 연관 패스웨이 발굴을 통해 질병에 대한 생물학적 기전을 해석하는데 기여할 수 있을 것으로 기대하고 있다. 이번 연구는 생물정보학 분야의 세계적인 저널인 Briefings in Bioinformatics 최근호에 게재되었다.
[연구결과]
DeepHisCoM: Deep learning pathway analysis using hierarchical structural component models
Chanwoo Park1, Boram Kim2 and Taesung Park1,2,*
1Department of Statistics, Seoul National University, Seoul 08826, Korea
2Interdisciplinary Program in Bioinformatics, Seoul National University, Seoul 08826, Korea
*Correspondence
유전자와 단백질과 같은 생물학적 인자는 서로간의 복잡한 상호작용, 즉 패스웨이를 통하여 질병에 영항을 끼치게 된다. 따라서 이러한 생물학적 인자의 패스웨이에 대한 복잡한 비선형적 영향을 반영하기 위하여 본 연구에서는 딥 러닝을 활용한 패스웨이 분석 방법론인 DeepHisCoM을 개발하였다.
DeepHisCoM의 성능을 확인하기 위하여 간암 환자의 대사체, 전사체, 그리고 메타지놈 자료에 모델을 적용하여 간암과 연관된 패스웨이를 성공적으로 찾았으며, 특히 COVID-19 환자의 중증도와 연관된 패스웨이를 찾기 위해 UK Biobank의 SNP 자료를 활용하여 MAPK signaling pathway, GnRH signaling pathway, hypertrophic cardiomyopathy, and dilated cardiomyopathy와 같은 기존에 COVID-19과 연관이 있는 것으로 밝혀진 다수의 패스웨이를 찾았다.
[용어설명]
SNP: DNA 염기서열에서 하나의 염기서열(A,T,G,C)의 차이를 보이는 유전적 변화 또는 변이를 단일 핵산염기 다형현상(Single Nucleotide polymorphism, SNP)이라고 하며, 스닙이라고 읽는다. 인구집단에서 1% 이상의 빈도로 존재하는 2개 이상의 대립 염기서열이 발생하는 위치를 SNP이라고 하며 대립유전자형이 5%이상의 빈도로 존재하는 경우 common polymorphism이라고 하며, 1~5%인 경우 rare polymorphism으로 분류한다.
[그림설명]
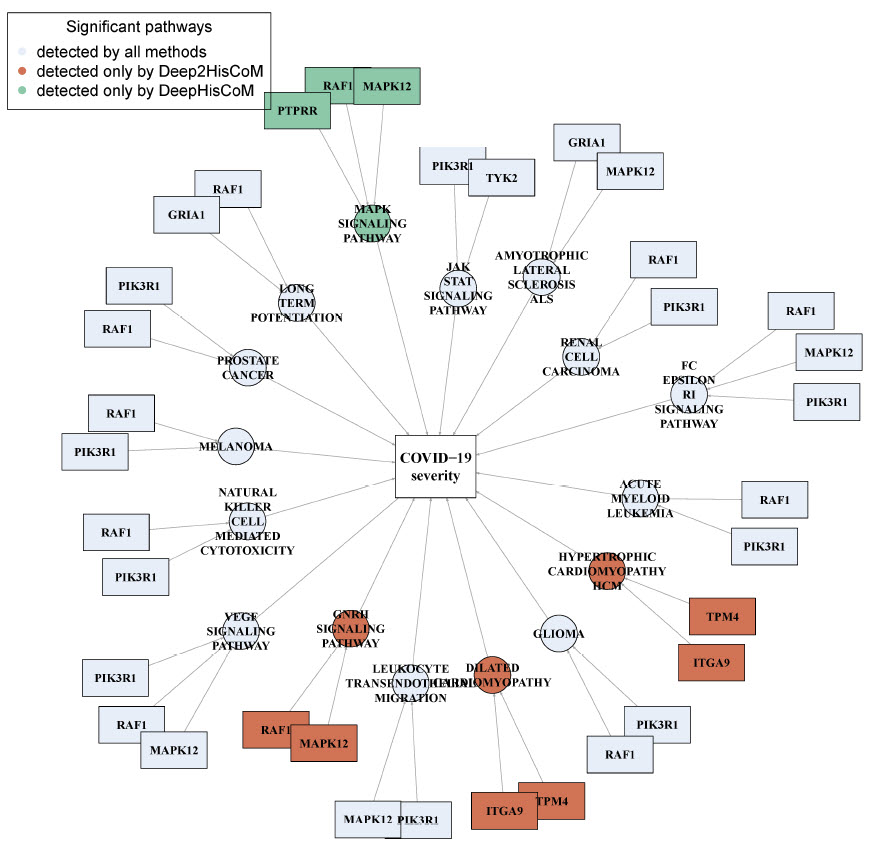
COVID-19 중증도 연관 패스웨이 및 유전자의 시각화
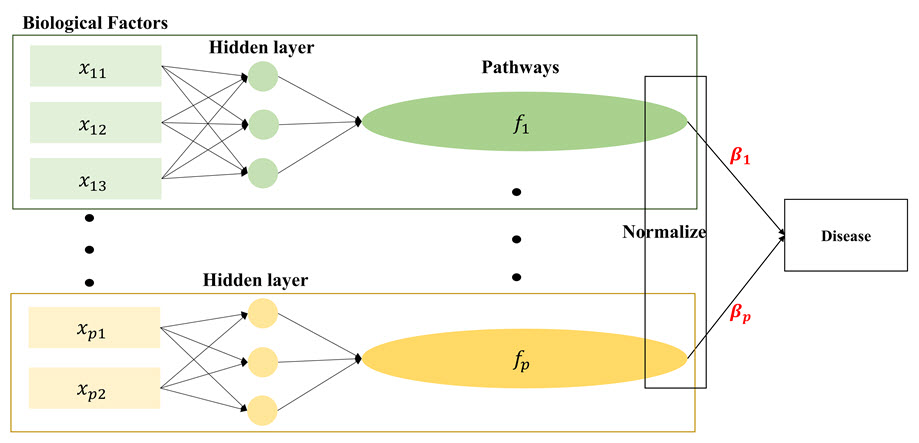
DeepHisCoM 모형의 구조. 직사각형은 바이오마커 원은 hidden layer, 그리고 타원은 패스웨이를 의미

