지방간질환의 유전형 기반 개인별 치료법 제시
[연구필요성]
지방간질환의 조기 진단과 진행 예측을 위한 바이오마커 개발, 치료제 개발은 아직까지 모두 제한적인 상황이다. 이러한 한계는 질환의 조기 발견과 효과적인 치료를 어렵게 하며, 환자의 건강을 위협하고 있다. 이러한 제약들의 가장 큰 원인 중 하나는 환자 개개인의 유전적 배경에 따라 질병의 발현과 진행 양상이 크게 달라지지만, 이에 기반한 예측 바이오마커 및 치료법은 아직 부족하다. 특히, 기존 연구는 서구 중심으로 진행되어 동아시아인을 포함한 다양한 인종을 대표하지 못하는 한계가 있었다.
[연구성과/기대효과]
이번 연구는 서울대학교 의과대학, 서울시보라매병원, 서울대병원, 서울대 약학대학, 한국생명공학연구원(KRIBB) 등 국내 연구진이 협력하여 간 조직을 단일 세포 수준으로 분석한 세계적인 수준의 유전체 연구이다. 연구진은 다양한 지방간질환 단계(단순 지방간, 경증 섬유화, 중증 섬유화 등)를 포괄하는 환자 30명과 건강 대조군 24명의 간 조직을 수집해 총 24만 개 이상의 세포를 분석하였다. 연구진은 최신 유전체 분석 기법인 단일세포 RNA 분석법(snRNA-seq)과 유전자 발현 연관 분석(eQTL)법을 결합하여 3,500개 이상의 유전자-유전자 발현 조절 변이 연관관계를 찾아냈고, 세포 상태에 따라 유전자 조절 방식이 달라지는 상호작용형 eQTL (interacting-eQTL)도 규명했다. 이 연구방법은 기존의 질환 조직 분석법에 환자 개인 특이적 유전형과 환자 조직 내 세포 특이적 상태를 구현하는 독창적인 유전체 데이터 분석 방법으로, 연구진은 이러한 방법으로 약 600개 정도의 환자 특이적, 세포 특이적 유전자 발현 조절 모듈을 발견하였다. 특히 그 중, FOXO1이라는 전사인자와 EFHD1 유전자 사이의 상호작용이 특정 유전형을 가진 일부의 환자들에게서 대사이상 지방간질환 악화에 핵심적으로 역할함을 제안하였고 이를 실험적으로 입증하였다. 질환 유전체 연구가 시작된 지 20년이 되어가는 시점에서 이 연구는 기존 연구 방법론을 뛰어넘는 새로운 유전자 분석법을 제안하여 한 걸음 더 인간 유전체의 복잡성과 환자 조직의 생리학적 복잡성을 재현해 낸 연구이며, 실제 지방간질환의 진행을 좌우하는 유전적 요인을 밝히고, 환자의 유전자에 맞춘 맞춤형 치료의 가능성을 제시한다는 점에서 큰 의의가 있다. 본 연구는 개별 환자의 유전형과 간 조직 내 세포 상태를 반영한 분석을 통해, 질환의 이질성과 맞춤형 치료 타겟 규명의 가능성을 보여주었다. 특히 MASLD의 유전적 기전을 세포 유형과 공간적 위치(예: periportal vs pericentral hepatocytes) 단위로 규명함으로써, 향후 유전형 기반 환자 선별 및 정밀치료에 이론적 근거를 제공한다. 이 접근은 향후 고혈압, 당뇨, 치매 등 다양한 만성질환에도 적용 가능하다. 본 연구는 한국연구재단과 보건산업진흥원 과제들의 연구지원을 받아 수행되었다.
[본문]
□ 질환 소개와 기존 연구의 한계점
대사이상 지방간질환(metabolic dysfunction-associated steatotic liver disease; MASLD, 이하 지방간질환)은 전 세계 성인의 약 25%가 앓고 있는 대표적인 만성 간질환으로, 유병률이 전 세계적으로 빠르게 증가하고 있다. 비만·당뇨병 등 대사 이상과 관련된 간 내 지방 축적이 특징으로 초기엔 증상이 없으나 간염, 간경변, 간암으로 진행될 수 있으며, 주요 사망 원인은 심혈관 질환이다. 체중 감량과 생활습관 개선이 예방과 치료 방안으로 중요하지만 다른 만성질환과 같이 개인마다 다양한 유전적인 소인도 중요한 인자로 작용하고 있다.
지방간질환의 조기 진단과 진행 예측을 위한 바이오마커 개발, 치료제 개발은 아직까지 모두 제한적인 상황이다. 이러한 한계는 질환의 조기 발견과 효과적인 치료를 어렵게 하며, 환자의 건강을 위협하고 있다. 이러한 제약들의 주요 원인 중 하나는 환자 개개인의 유전적 배경에 따라 질병의 발현과 진행 양상이 크게 달라지지만, 이에 기반한 예측 바이오마커 및 치료법은 아직 부족하다. 특히, 기존 연구는 서구 중심으로 진행되어 동아시아인을 포함한 다양한 인종을 대표하지 못하는 한계가 있었다.
□ 본 연구의 내용과 성과
이번 연구는 서울대학교 의과대학, 서울특별시보라매병원, 서울대학교병원, 서울대학교 약학대학, 한국생명공학연구원(KRIBB) 등 국내 연구진이 협력하여 간 조직을 단일 세포 수준으로 분석한 세계적인 수준의 유전체 연구이다. 연구진은 다양한 지방간질환 단계(단순 지방간, 경증 섬유화, 중증 섬유화 등)를 포괄하는 환자 30명과 건강 대조군 24명의 간 조직을 수집해 총 24만 개 이상의 세포를 분석하였다.
연구진은 최신 유전체 분석 기법인 단일세포 RNA 분석법(snRNA-seq)과 유전자 발현 연관 분석(eQTL)법을 결합하여 3,500개 이상의 유전자-유전자 발현 조절 변이 연관관계를 찾아냈고, 세포 상태에 따라 유전자 조절 방식이 달라지는 상호작용형 eQTL (interacting-eQTL)도 규명했다. 이 연구 방법은 기존의 질환 조직 분석법에 환자 개인 특이적 유전형과 환자 조직 내 세포 특이적 상태를 구현하는 독창적인 유전체 데이터 분석 방법으로, 연구진은 이러한 방법으로 약 600개 정도의 환자 특이적, 세포 특이적 유전자 발현 조절 모듈을 발견하였다. 특히 그 중, FOXO1이라는 전사인자와 EFHD1 유전자 사이의 상호작용이 특정 유전형을 가진 일부의 환자들에게서 대사이상 지방간질환 악화에 핵심적으로 역할함을 제안하였고 이를 실험적으로 입증하였다.
질환 유전체 연구가 시작된 지 20년이 되어가고 있는 시점에서 이 연구는 기존의 연구 방법론을 뛰어넘는 새로운 유전자 분석법을 제안하여 한 걸음 더 인간 유전체의 복잡성과 환자 조직의 생리학적 복잡성을 재현해 낸 연구이며, 실제 지방간질환의 진행을 좌우하는 유전적 요인을 밝히고, 환자의 유전자에 맞춘 맞춤형 치료의 가능성을 제시한다는 점에서 큰 의의가 있다.
본 연구는 개별 환자의 유전형과 간 조직 내 세포 상태를 반영한 분석을 통해, 질환의 이질성과 맞춤형 치료 타겟 규명의 가능성을 보여주었다. 특히 MASLD의 유전적 기전을 세포 유형과 공간적 위치(예: periportal vs pericentral hepatocytes) 단위로 규명함으로써, 향후 유전형 기반 환자 선별 및 정밀치료에 이론적 근거를 제공한다. 이 접근은 향후 고혈압, 당뇨, 치매 등 다양한 만성질환에도 적용 가능하다. 본 연구는 한국연구재단과 보건산업진흥원 과제들의 연구 지원을 받아 수행되었다.
[연구결과]
※ 본 연구는 유전학 분야 최고 저널인 Nature Genetics에 게재되었다.
Single-cell eQTL analysis identifies genetic variation underlying metabolic dysfunction-associated steatohepatitis
Sung Eun Hong, Seon Ju Mun, Young Joo Lee, Taekyeong Yoo, Kyung-Suk Suh, Keon Wook Kang, Myung Jin Son, Won Kim & Murim Choi
(Nature Genetics, https://www.nature.com/articles/s41588-025-02237-8)
- ○eQTL : expression quantitative trait loci, 특정 유전자의 발현을 직접적으로 조절하는 유전 변이들
[그림설명]
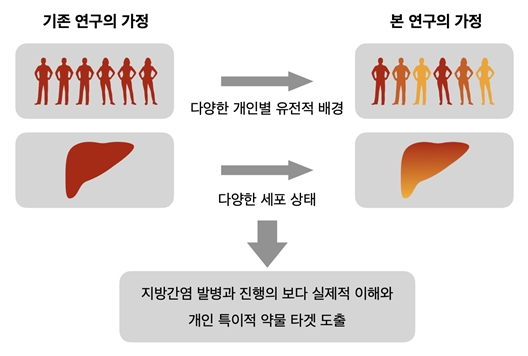
그림 1. 전체적인 연구 가설과 목표. 개인별 다양한 유전적 배경과 각 환자가 보유한 간 조직 내에서의 다양한 세포 상태를 실험적으로 구현하여 분석을 진행함
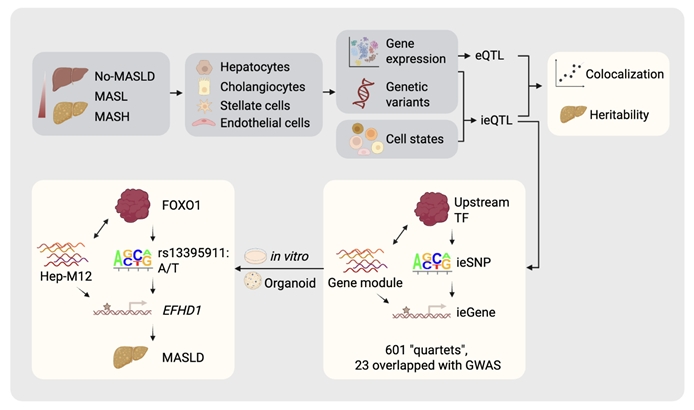
그림 2. 전체적인 연구의 흐름과 결과. 단일세포전사체 데이터와 전장유전체분석법을 혼합하는 eQTL 방법을 지방간질환 환자와 대조군에 적용하여 분석과 실험을 진행하였음



